
2025 年 10 月,《Hierarchical affinity landscape navigation through learning a shared pocket-ligand space》作为封面文章发表于《Patterns》(三大顶刊之一《Cell》旗下期刊),发布名为 LigUnity 的亲和力基础模型,该模型可在极低实验成本下快速发现高潜力分子,在保证高精度的同时,早筛速度较传统方法提升超万倍,且支持自主探索全新药物靶点,新药开发实现跨数量级降本增效。
该研究由粤港澳大湾区数字经济研究院(IDEA)AI4Science 团队、晶泰科技(XtalPi)及华盛顿大学的研究团队联合完成。
传统药物发现流程往往需要数年时间、数亿美元成本,过程犹如大海捞针:首先需从数亿分子中,初筛出少数可能有效的“苗头化合物”,随后需合成并测试数百上千个衍生化合物,对这些分子进行耗时漫长的“打磨”与“优化”。
LigUnity 通过构建蛋白质与配体的共享表示空间,让 AI 统一学习并驾驭从大规模筛选到精细活性优化的完整药物发现流程,智能化高效并行筛选和优化,任务处理效率达到分钟级,极大程度地压缩了药物早筛与优化周期。同时,凭借强大的泛化能力,LigUnity 能够自主探索全新药物靶点,为开拓前沿治疗领域提供重要技术底座。
在全球医药研发数字化和智能化加速演进的背景下,LigUnity 的推出为制药产业带来从“实验主导”向“模型主导”的转型支点,使 AI 在药物发现领域由任务模型向基础模型跃进,使药物研发能够在统一框架下共享模型能力与数据生态,推动 AI 成为新药发现的核心生产力,为 AI4Science 从单点突破走向平台化的系统协作奠定了技术基础。IDEA AI4Science 团队也将在即将到来的 IDEA 大会上首发 AI 自主驱动的药物发现平台,进一步推动科学研究领域的智能赋能。
LigUnity
下一代 AI 驱动药物发现的基础模型
从“数周”到“分钟”
药物早筛速度提升超万倍
在人体中,上万种蛋白质维持着复杂的生命活动,其中许多蛋白质的异常与疾病直接相关,是潜在的药物靶点。然而,目前仅有约 10% 的蛋白质能被已知的药物分子高效结合,绝大多数靶点仍处于“黑暗” 之中,等待着被有效的药物“点亮”,难度相当于通过望远镜在银河宇宙中寻找新星。
药物研发中,为这些靶点找到合适的药物分子,有两个关键步骤和关键要求:
传统方法中,整个过程一般耗时数年,且需要使用不同的工具和模型,这种工具的割裂也导致模型在筛选和优化上的效果受到限制:专注于筛选的模型可能无法精确区分结构相似但活性差异巨大的分子,专注于优化的模型则难以泛化到具有全新化学骨架的潜力分子。
LigUnity 创新地通过统一的亲和力基础模型,让 AI 学会一种能同时理解蛋白质结合口袋和药物分子的“共同语言”,使苗头化合物发现与优化两个步骤可通过同一模型完成,数分钟内即可完成筛选和优化,也使探索更为广阔、结构新颖的化学空间成为可能。
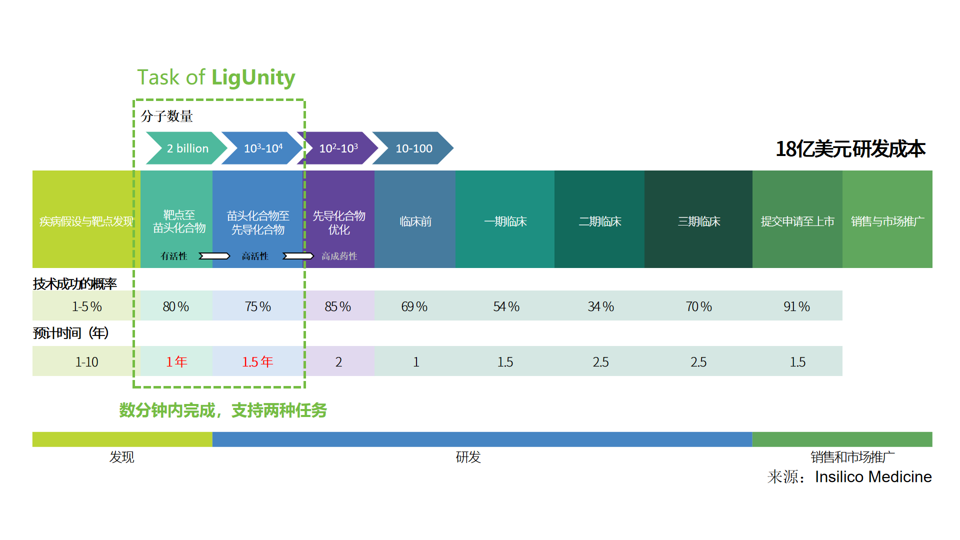
这依托于研究团队构建的迄今为止规模最大的、以实验(Assay)组织的亲和力数据库 PocketAffDB,其中包含了蛋白质口袋结构信息和近百万活性数据。基于此,LigUnity 通过一种创新的“层级式学习”策略进行训练:
在预训练期间,LigUnity 同时优化上述目标;在推理时,LigUnity 将蛋白和口袋映射至同一空间,使用余弦相似度计算其活性。在保证了高精度的同时,LigUnity 的速度比传统的分子对接方法(如 Glide-SP)快万倍。

卓越的泛化能力
攻克“无药靶点”
药物发现最核心的挑战在于应对未知。对于全新靶点或全新化学骨架,模型的泛化能力是其能否投入实际应用的关键。
LigUnity 在严格的“零样本”与“小样本”设定下,对全新蛋白质靶点和全新分子骨架均展现出卓越的预测能力。这得益于其从海量结构-活性数据中学习并形成对蛋白-配体相互作用底层规律的深刻理解。
基于强大的泛化能力,LigUnity 能够直接应用于缺乏活性分子、甚至是在传统上被认为“不可成药”的靶点探索中。它极大地降低了项目启动的门槛,也为研发团队挑战前沿生物学、开拓全新治疗领域提供了技术支撑。
实验结果
虚拟筛选任务
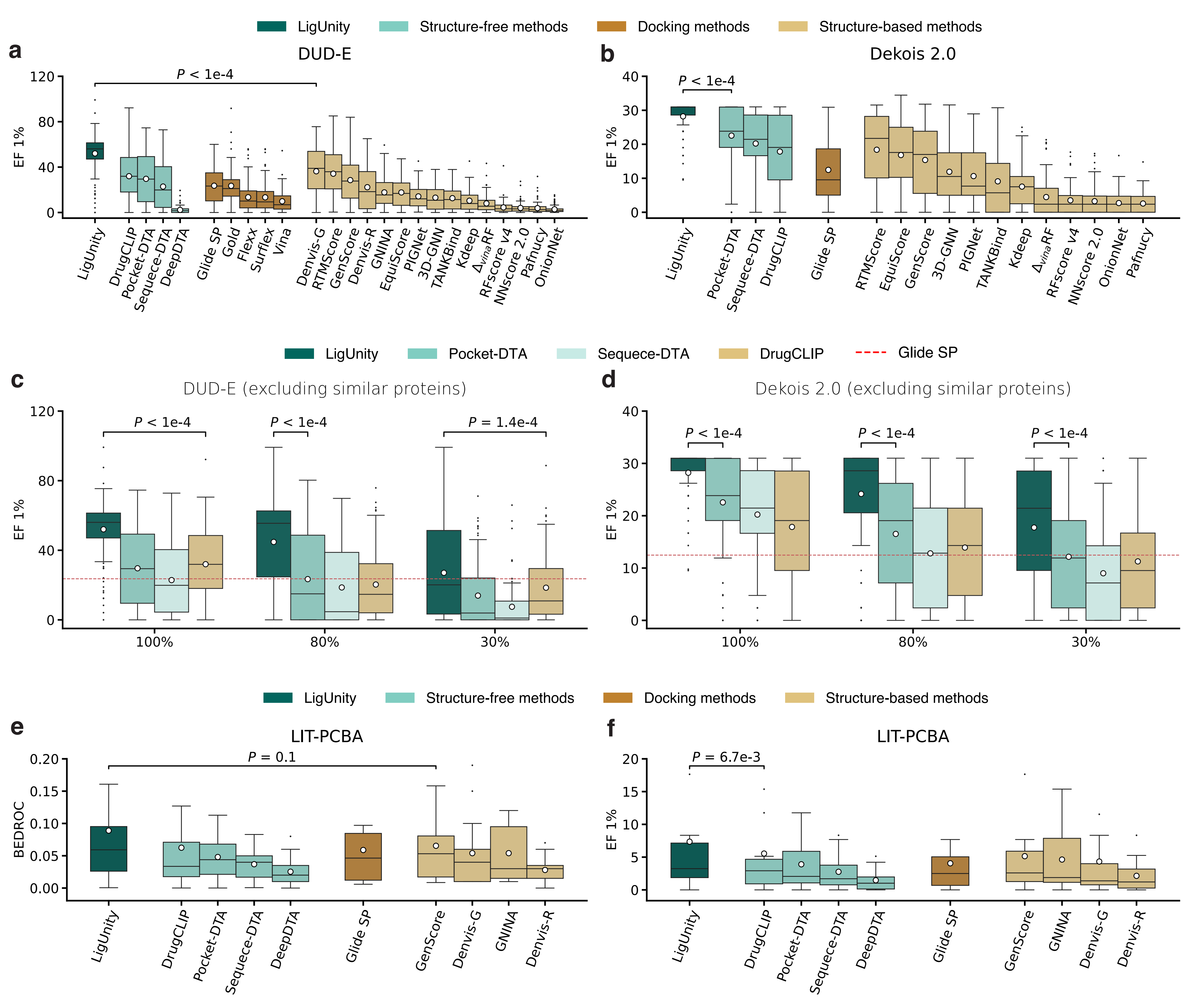
全面领先:在 DUD-E、DEKOIS 2.0 和 LIT-PCBA 这三个主流虚拟筛选基准测试中,LigUnity 的表现超越了全部 24 种竞争方法,包括传统的分子对接软件和各类机器学习模型。
性能巨大提升:与表现最好的基线模型相比,LigUnity 在关键指标“富集因子 (EF 1%)”上取得了超过 50% 的性能提升(p < 10⁻⁹)。
强大的泛化能力:即便在排除了与训练集中蛋白质相似(序列相似性 < 30%)的新靶点上进行测试,LigUnity 的性能依然显著优于 Glide SP,表现出对未知靶点的强大预测能力和实用价值。
苗头化合物优化任务
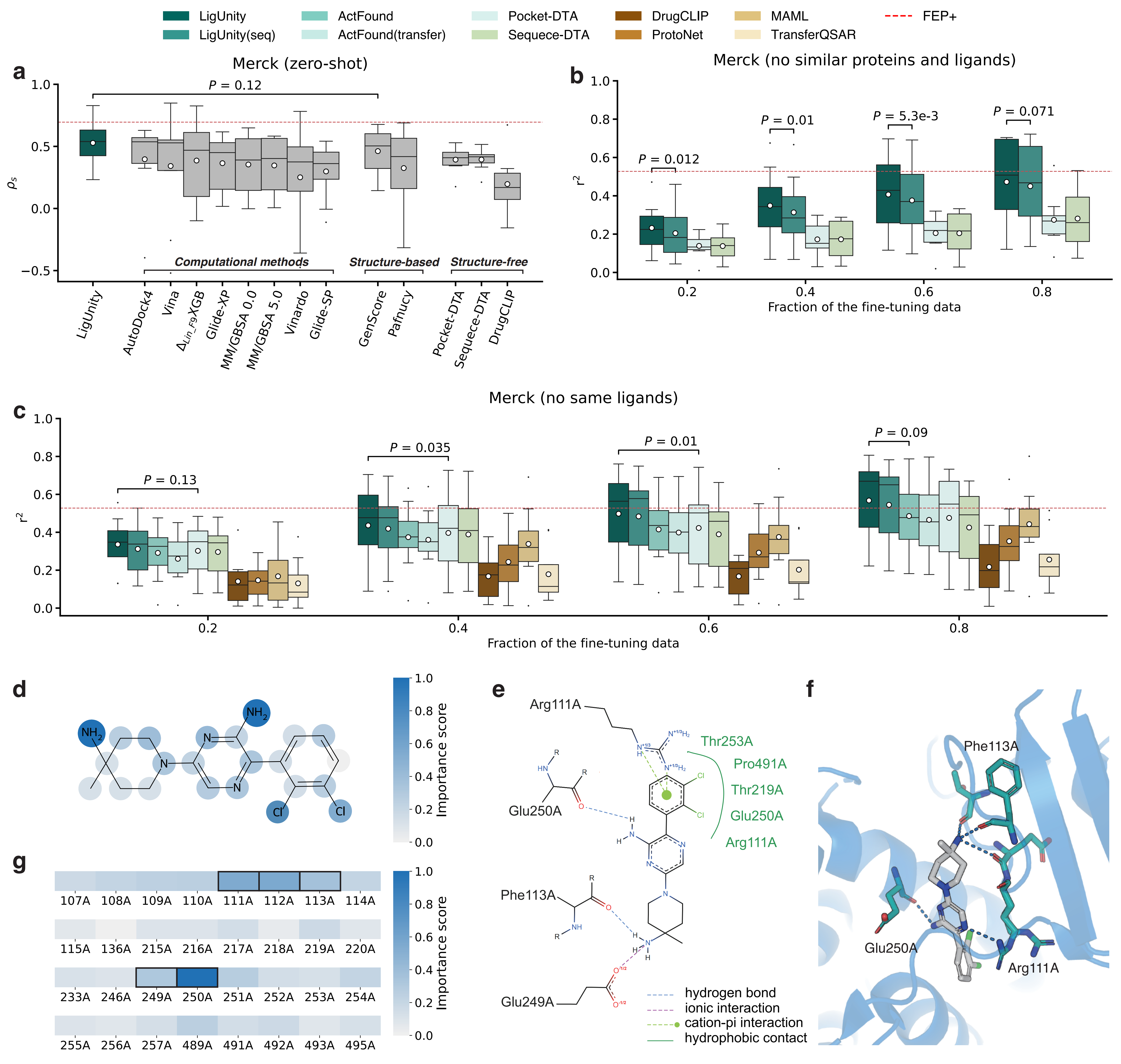
零样本性能优越:在 JACS 和 Merck 这两个行业公认的 FEP 基准测试中,在不使用任何目标数据(zero-shot)的情况下,LigUnity 的预测性能已优于 Glide-SP、MM/GBSA 等传统计算方法及其他机器学习模型,展现了其强大的预训练知识。
少量数据即可媲美“金标准”:当使用少量(10-20 个)已知数据进行微调后,LigUnity 的预测精度(在 Merck 基准上 r² = 0.472)已接近计算成本极度高昂的物理计算「金标准」方法 FEP+(r² = 0.528),为昂贵的实验和计算提供了高性价比的替代方案。
良好的可解释性:尽管未使用蛋白 – 分子结合构象作为输入,LigUnity 仍成功识别出了对结合起关键作用的配体原子和口袋残基,与已知的晶体结构相互作用模式高度吻合,为化学家优化分子提供了可靠指导。
多场景应用的通用性
前瞻研究与骨架跃迁:在模拟真实药物发现的测试中,LigUnity 表现出众。在时间分割(用历史数据预测未来新分子)和化学骨架分割(泛化至训练集中未见的全新化学骨架)设定下,它均超越了所有基线模型,证明其具备突破现有化学空间、发现新潜力分子的强大泛化能力。
对多源、异构数据的鲁棒性:真实世界的生物活性数据来源多样、测量单位不一(如 nM vs. % 抑制率)。得益于其独特的“列表排序”机制,LigUnity 天然对测量单位不敏感。在 OOD 测试中,其性能相比传统回归模型提升高达 40.2%,展现了其作为基础模型处理复杂、真实世界数据的卓越适应性。
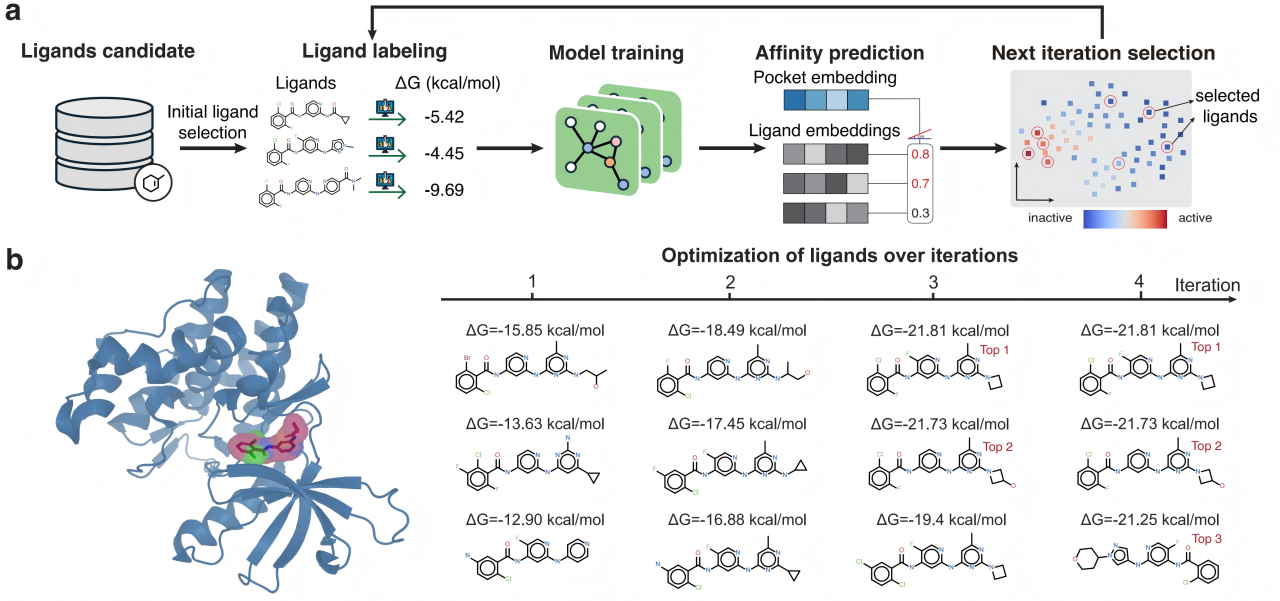
主动学习应用:在模拟真实研发流程的主动学习框架中,研究团队对 TYK2 靶点进行研究,从 10,000 个后续分子中找到活性最好的分子。集成 LigUnity 的框架仅通过 4 轮迭代(400 活性标注数据),就成功找到了活性最高的 3 个分子,证明其能以极少的实验成本,高效地指导药物发现进程。

LigUnity 通过一个统一的、层级式的学习框架,成功地将虚拟筛选与活性优化相结合,在速度、精度和泛化性上均取得了突破。它不仅是一个强大的预测工具,更是一个具备广泛适用性的基础模型,为加速早期药物发现的全流程奠定了坚实的基础。

2025 年 10 月,《Hierarchical affinity landscape navigation through learning a shared pocket-ligand space》作为封面文章发表于《Patterns》(三大顶刊之一《Cell》旗下期刊),发布名为 LigUnity 的亲和力基础模型,该模型可在极低实验成本下快速发现高潜力分子,在保证高精度的同时,早筛速度较传统方法提升超万倍,且支持自主探索全新药物靶点,新药开发实现跨数量级降本增效。
该研究由粤港澳大湾区数字经济研究院(IDEA)AI4Science 团队、晶泰科技(XtalPi)及华盛顿大学的研究团队联合完成。
传统药物发现流程往往需要数年时间、数亿美元成本,过程犹如大海捞针:首先需从数亿分子中,初筛出少数可能有效的“苗头化合物”,随后需合成并测试数百上千个衍生化合物,对这些分子进行耗时漫长的“打磨”与“优化”。
LigUnity 通过构建蛋白质与配体的共享表示空间,让 AI 统一学习并驾驭从大规模筛选到精细活性优化的完整药物发现流程,智能化高效并行筛选和优化,任务处理效率达到分钟级,极大程度地压缩了药物早筛与优化周期。同时,凭借强大的泛化能力,LigUnity 能够自主探索全新药物靶点,为开拓前沿治疗领域提供重要技术底座。
在全球医药研发数字化和智能化加速演进的背景下,LigUnity 的推出为制药产业带来从“实验主导”向“模型主导”的转型支点,使 AI 在药物发现领域由任务模型向基础模型跃进,使药物研发能够在统一框架下共享模型能力与数据生态,推动 AI 成为新药发现的核心生产力,为 AI4Science 从单点突破走向平台化的系统协作奠定了技术基础。IDEA AI4Science 团队也将在即将到来的 IDEA 大会上首发 AI 自主驱动的药物发现平台,进一步推动科学研究领域的智能赋能。
LigUnity
下一代 AI 驱动药物发现的基础模型
从“数周”到“分钟”
药物早筛速度提升超万倍
在人体中,上万种蛋白质维持着复杂的生命活动,其中许多蛋白质的异常与疾病直接相关,是潜在的药物靶点。然而,目前仅有约 10% 的蛋白质能被已知的药物分子高效结合,绝大多数靶点仍处于“黑暗” 之中,等待着被有效的药物“点亮”,难度相当于通过望远镜在银河宇宙中寻找新星。
药物研发中,为这些靶点找到合适的药物分子,有两个关键步骤和关键要求:
传统方法中,整个过程一般耗时数年,且需要使用不同的工具和模型,这种工具的割裂也导致模型在筛选和优化上的效果受到限制:专注于筛选的模型可能无法精确区分结构相似但活性差异巨大的分子,专注于优化的模型则难以泛化到具有全新化学骨架的潜力分子。
LigUnity 创新地通过统一的亲和力基础模型,让 AI 学会一种能同时理解蛋白质结合口袋和药物分子的“共同语言”,使苗头化合物发现与优化两个步骤可通过同一模型完成,数分钟内即可完成筛选和优化,也使探索更为广阔、结构新颖的化学空间成为可能。

这依托于研究团队构建的迄今为止规模最大的、以实验(Assay)组织的亲和力数据库 PocketAffDB,其中包含了蛋白质口袋结构信息和近百万活性数据。基于此,LigUnity 通过一种创新的“层级式学习”策略进行训练:
在预训练期间,LigUnity 同时优化上述目标;在推理时,LigUnity 将蛋白和口袋映射至同一空间,使用余弦相似度计算其活性。在保证了高精度的同时,LigUnity 的速度比传统的分子对接方法(如 Glide-SP)快万倍。

卓越的泛化能力
攻克“无药靶点”
药物发现最核心的挑战在于应对未知。对于全新靶点或全新化学骨架,模型的泛化能力是其能否投入实际应用的关键。
LigUnity 在严格的“零样本”与“小样本”设定下,对全新蛋白质靶点和全新分子骨架均展现出卓越的预测能力。这得益于其从海量结构-活性数据中学习并形成对蛋白-配体相互作用底层规律的深刻理解。
基于强大的泛化能力,LigUnity 能够直接应用于缺乏活性分子、甚至是在传统上被认为“不可成药”的靶点探索中。它极大地降低了项目启动的门槛,也为研发团队挑战前沿生物学、开拓全新治疗领域提供了技术支撑。
实验结果
虚拟筛选任务
全面领先:在 DUD-E、DEKOIS 2.0 和 LIT-PCBA 这三个主流虚拟筛选基准测试中,LigUnity 的表现超越了全部 24 种竞争方法,包括传统的分子对接软件和各类机器学习模型。
性能巨大提升:与表现最好的基线模型相比,LigUnity 在关键指标“富集因子 (EF 1%)”上取得了超过 50% 的性能提升(p < 10⁻⁹)。
强大的泛化能力:即便在排除了与训练集中蛋白质相似(序列相似性 < 30%)的新靶点上进行测试,LigUnity 的性能依然显著优于 Glide SP,表现出对未知靶点的强大预测能力和实用价值。

苗头化合物优化任务
零样本性能优越:在 JACS 和 Merck 这两个行业公认的 FEP 基准测试中,在不使用任何目标数据(zero-shot)的情况下,LigUnity 的预测性能已优于 Glide-SP、MM/GBSA 等传统计算方法及其他机器学习模型,展现了其强大的预训练知识。
少量数据即可媲美“金标准”:当使用少量(10-20 个)已知数据进行微调后,LigUnity 的预测精度(在 Merck 基准上 r² = 0.472)已接近计算成本极度高昂的物理计算「金标准」方法 FEP+(r² = 0.528),为昂贵的实验和计算提供了高性价比的替代方案。
良好的可解释性:尽管未使用蛋白 – 分子结合构象作为输入,LigUnity 仍成功识别出了对结合起关键作用的配体原子和口袋残基,与已知的晶体结构相互作用模式高度吻合,为化学家优化分子提供了可靠指导。

多场景应用的通用性
前瞻研究与骨架跃迁:在模拟真实药物发现的测试中,LigUnity 表现出众。在时间分割(用历史数据预测未来新分子)和化学骨架分割(泛化至训练集中未见的全新化学骨架)设定下,它均超越了所有基线模型,证明其具备突破现有化学空间、发现新潜力分子的强大泛化能力。
对多源、异构数据的鲁棒性:真实世界的生物活性数据来源多样、测量单位不一(如 nM vs. % 抑制率)。得益于其独特的“列表排序”机制,LigUnity 天然对测量单位不敏感。在 OOD 测试中,其性能相比传统回归模型提升高达 40.2%,展现了其作为基础模型处理复杂、真实世界数据的卓越适应性。
主动学习应用:在模拟真实研发流程的主动学习框架中,研究团队对 TYK2 靶点进行研究,从 10,000 个后续分子中找到活性最好的分子。集成 LigUnity 的框架仅通过 4 轮迭代(400 活性标注数据),就成功找到了活性最高的 3 个分子,证明其能以极少的实验成本,高效地指导药物发现进程。


LigUnity 通过一个统一的、层级式的学习框架,成功地将虚拟筛选与活性优化相结合,在速度、精度和泛化性上均取得了突破。它不仅是一个强大的预测工具,更是一个具备广泛适用性的基础模型,为加速早期药物发现的全流程奠定了坚实的基础。
